|
 Die
Firma Promega
hat einen Baukasten
zum genetischer Fingerabdruck entwickelt, wir haben ihn ausprobiert.
Die
Firma Promega
hat einen Baukasten
zum genetischer Fingerabdruck entwickelt, wir haben ihn ausprobiert.
Der genetische
Fingerabdruck - ein Projekt im Rahmen der Biotechnologie an der
Max-Beckmann-Schule in Kooperation mit Promega.
DNA-Detektive
- Simulation der forensischen DNA-Analyse
Im
Versuch wird durch DNA-Analyse ein fiktives Verbrechen aufgeklärt. Als
Ausgangsmaterial dienen DNA-Proben von mehreren fiktiven
Verdachtspersonen. Eine davon ist der Täter und hat am Tatort eine
DNA-Spur zurückgelassen. Diese DNA wird in der Praxis aus biologischem
Material wie Speichel, Sperma oder Blut gewonnen. Bei unserem Versuch wird
sie aus der Mundschleimhaut gewonnen. Zwei elementare Techniken des
"genetischen Fingerprintings" werden gezeigt. Die exponentielle
Vermehrung (Amplifikation) der DNA mit der Polymerase Kettenreaktion (PCR)
und die Auftrennung der entstehenden DNA-Bruchstücke mittels
Agarose-Gelelektrophorese.
Im
Ergebnis wird eine Bandenmuster der Verdächtigen erhalten - auch
genetischer Fingerabdruck genannt - der mit der Tatort-DNA verglichen
wird. Stimmen die Muster überein, wurde der Täter ermittelt. Die Methode
des "genetischen Fingerprints" wird wegen ihrer hohen
Beweiskraft besonders bei der Vaterschaftsbestimmung und der Aufklärung
von Straftaten genutzt.
Wie
funktioniert es? - Die
Mikrosatelliten-DNA
- »short
tandem repeats«
Das
Genom eines jeden Menschen ist einmalig (von eineiigen Zwillingen einmal
abgesehen). Die genetischen Unterschiede sind aber recht gering, besonders
wenn es sich um wichtige Gene handelt. Es gibt aber Abschnitte auf den
menschlichen Chromosomen, die bei allen Menschen verschieden sind. Diese
Abschnitte enthalten die Mikrosatelliten-DNA, die aus kurzen (circa 5
Basenpaare (bp)) Basensequenzen besteht, die sich mehrmals nacheinander
wiederholen. Diese im englischen Sprachgebrauch »short tandem repeats« (STR)
genannten Regionen können leicht aus dem Gesamterbmaterial gewonnen
werden und sind wie ein Fingerabdruck. Das Ergebnis dieser Analyse nach
der PCR und Elektrophorese ist ein Strichmuster , das von der Abfolge der
Basen und individuellen Wiederholungen in der Mikrosatelliten-DNA abhängt:
der genetische Fingerabdruck. Dieses Muster wird nach den Mendelschen
Regeln vererbt. Das Muster eines Kindes setzt sich aus dem mütterlichen
und dem väterlichen Muster zusammen, so dass zumindest einige Banden
mit denen des Vaters übereinstimmen müssen, für einen Menschen
ist es aber, vorausgesetzt man untersucht mehrere Genorte (Loci),
einzigartig. Die STR (short
tandem repeats) beruhen darauf, dass verschiedene Individuen einer
Population an bestimmten Stellen im Genom unterschiedlich viele
tandemartige wiederholte, 3
bis 7 bp lange, Sequenzen
besitzen.
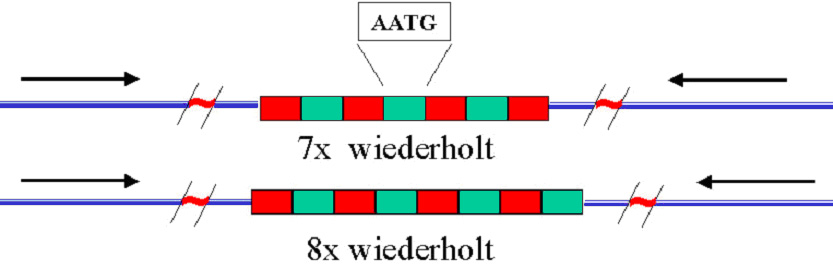
So
kann die Sequenz AATG (TPOX-Locus) bei einer oder acht mal wiederholt
vorliegen (Siehe Abbildung).
Diese
häufig vorkommenden Wiederholungen (repeats) sind über das
gesamte menschliche Genom verteilt und eine somit reichhaltige Quelle hoch
polymorpher und durch PCR zu detektierende Marker. Dadurch
lassen sich mehr oder weniger alle Individuen einer Population bzgl.
dieser Sequenzen voneinander unterscheiden, weswegen sich STRs sehr gut für
die Unterscheidung beliebiger Individuen einer Population eignen.
So kann die Sequenz AATG (TPOX-Locus)
bei einer oder acht mal wiederholt vorliegen (siehe Abbildung).
Durch
Vergleich mit Markern können die Größen der einzelnen mit der
Elektrophorese getrennten Fragmente bestimmt werden:
pGEM®
Marker
Die pGEM® DNA Marker sind sichtbare Standards mit denen die Größenbereiche
der Allele für die Loci bestimmt werden können. Die Marker bestehen aus
15 DNA-Fragmenten mit den folgenden Größen:
Die Wiederholungen für die einzeln Loci liegen in einem Größenbereich
von:
Basenpaare:
2645, 1605, 1198, 676, 517, 460, 396 ,350, 222, 179, 126, 75, 65, 51, 36
Bei
unseren Versuchen wurden die drei Genorte CSF1PO-
TPOX-
und THO1-Locus,
untersucht. Die Größen der
einzelnen Fragmente sind:
295
und 327 bp (CSF1PO-Locus)
Human c-fms proto-oncogene for CSF-1 receptor gene
224
und 252 bp (TPOX-Locus)
Human thyroid peroxidase gene
179
und 203 bp (THO1-Locus)
Human thyrosine hydroxylase gene
STR-Tabelle:
| STR-Locus |
Chromosomaler
Locus (Genort) |
Größen
der
Allel-Leiter
(in bp)
|
Anzahl
der möglichen Wiederholungen für Kaukasier
|
Anzahl
der Wiederholungen in der K562 DNA (Positivkontrolle) |
Sequenzmotiv
derWiederholung
5’
®
3’
|
| CSF1PO |
Chromosom
5
(5q33.3-34)
|
295
- 327 |
7;
8; 9; 10; 11; 12; 13; 14; 15
78,1%
sind heterozygot (Kaukasier)
|
10;
9 |
AGAT |
| TPOX |
Chromosom
2
(2p25.1-pter)
|
224
- 252 |
6;
7;8; 9; 10; 11; 12; 13
64,7%
sind heterozygot (Kaukasier)
|
9;
8 |
AATG |
| THO1 |
Chromosom
11
(11p15.5)
|
179
- 203 |
5;
6; 7; 8; 9; 10; 11
76,5%
sind heterozygot (Kaukasier)
|
9,3;
9,3 |
AATG |
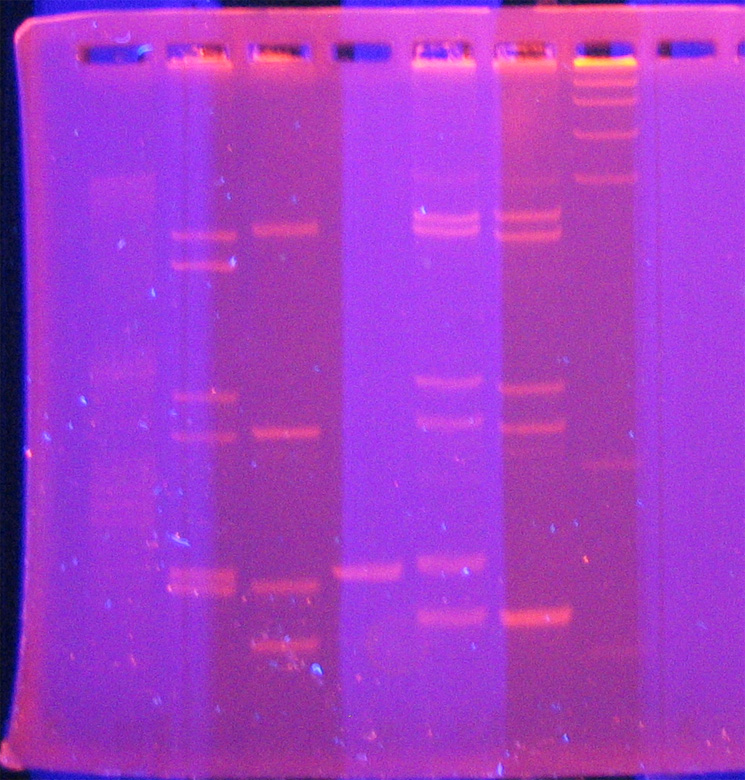
Unser Ergebnis mit einer
Digitalkamera aufgenommen: Spur 1 ist die Alleleleiter, die leider nicht
sehr gut aufgelöst wurde. Spur 2, 3, 5 und 6 sind Proben von Schülern.
Man sieht gut die Individualität, bei Spur 2,5 und 6 sind die
CSF1PO und TPOX loci im Gegensatz zu 3 homozygot . Spur 4 ist der Marker für
den THO1 Locus und Spur 7 ist der pGEM Marker.
|
 Die
Firma
Die
Firma 
